Był późny piątkowy wieczór wiosną 1983 r. Prawie pusta droga nr 128 z Berkeley do Anderson Valley. Zwykły początek kalifornijskiego weekendu. Nic nie wskazywało na to, że wydarzy się coś szczególnie ważnego, mogącego zmienić bieg historii badań genetycznych. Tego dnia Kary Mullis, jeden z szeregowych pracowników badawczych korporacji Cetus, jechał wraz ze swoją dziewczyną Jennifer Barnett na romantyczny weekend poza miastem. Jennifer spała, a Kary z braku innej rozrywki skupił się na poszukiwaniu odpowiedzi na fundamentalne pytanie dręczące od czasu do czasu każdego z nas: “co by było gdyby”. Większość ludzi w takiej sytuacji zadaje sobie istotne, życiowe pytania, w stylu: co by było gdyby wymienić Jennifer na jej seksowną blond koleżankę Andy. Na szczęście dla rozwoju genetyki oraz przypuszczalnie również dla Jennifer, tym razem Kary zastanawiał się nad zupełnie innym problemem. W biotechnologicznej firmie Cetus, Kary zajmował się pracą badawczą dotyczącą krótkich, jednoniciowych fragmentów DNA, nazywanych oligonukleotydami. Pytanie, które nurtowało Kary’ego tego wieczoru brzmiało: co by było gdyby w trakcie reakcji kopiowania DNA, dodać nie jeden, ale dwa oligonukleotydy. A co jeszcze ważniejsze, co by było gdyby sekwencje DNA dodanych oligonukleotydów, były tak zaprojektowane by móc przyłączyć się z obu stron kopiowanego fragmentu DNA. Dylemat z pozoru niezbyt istotny. Coś w rodzaju fundamentalnego pytania: czy wypić, jedno czy dwa piwa. Zazwyczaj i tak kończy się na ośmiu i pół oraz porannym bólu głowy. Na szczęście Kary będąc kierowcą pić chwilowo nie mógł i dzięki temu doprowadził swój przewód myślowy do końca. Zgodnie z relacją samego Kary’ego, gdy uświadomił sobie konsekwencje dodania dwóch nukleotydów do reakcji kopiowania DNA, doznał olśnienia. W analogicznej sytuacji ponad dwa tysiące lat wcześniej Archimedes wyskoczył z wanny krzycząc EUREKA!!! i wybiegł nago na ulicę by podzielić się swoim odkryciem z trochę zszokowanymi mieszkańcami Syrakuz. Kary zachował się równie impulsywnie, jednak tym razem obyło bez biegania nago po poboczu drogi nr 128. Zatrzymał samochód i krzycząc niezbyt składnie, starał się obudzić i przekonać Jennifer, że właśnie rozwiązał najbardziej denerwujący problem biochemii DNA. Choć był to faktycznie jeden z najważniejszych punktów zwrotnych historii badań DNA, Jennifer nie dała się obudzić. Spała dalej, nie zdając sobie sprawy, że właśnie traci szansę bycia naocznym świadkiem wielkiego odkrycia. Całą historię znamy ze szczegółami, dzięki bezpośredniej relacji Kary’ego, który opisał wszystkie zdarzenia tamtego wieczoru w swojej autobiografii pt. “Tańcząc nago na polu umysłu”. Jak widać towarzyszący nagłym, genialnym olśnieniom występ nago jednak się odbył. W przypadku Kary’ego tradycja ta została jednak spełniona wyłącznie w literackiej formie.
 Wnioski wyciągnięte przez Kary’ego Mullisa, podczas jazdy z Berkeley do Anderson Valley, okazały całkowicie prawidłowe. Dotyczyło to zarówno samego odkrycia jak i jego wagi oraz wpływu na rozwój badań genetycznych. Metoda opracowana przez Kary’ego stała się dla biologów molekularnych, tym czym było dla mikrobiologów udoskonalenie w XVII wieku mikroskopu przez Antoni van Leeuwenhoeka. Mikroskop pozwolił po raz pierwszy powiększyć i zbadać świat mikroorganizmów niewidocznych gołym okiem. Analogicznie metoda odkryta przez Kary’ego Mullisa pozwoliła na powiększenie liczby kopii dowolnego fragmentu DNA, do poziomu pozwalającego na swobodne badanie i odczytywanie informacji genetycznej. Metodzie opracowanej przez Kary’ego, nadano dość groźnie brzmiącą nazwę: Reakcja Łańcuchowa Polimerazy (Polymerase Chain Reaction), w skrócie PCR. Wbrew oczywistym skojarzeniom PCR nie ma nic wspólnego, z groźnym grzybem atomowym i tym czym zazwyczaj reakcja łańcuchowa kończy się w filmach z Bondem lub w Mission Impossible. W tym przypadku jest to zwykła zbieżność pojęć. Reakcja PCR, naśladuje naturalny proces kopiowania – replikacji DNA, pozwalając na zwielokrotnianie wybranego fragmentu informacji genetycznej (więcej na temat replikacji DNA znajdziecie we wpisie pt. “Genetyczny odcisk palca”). Zarówno w procesie naturalnej replikacji jak i w przypadku reakcji PCR enzymem odpowiedzialnym za szybkie i dokładne kopiowanie DNA jest polimeraza DNA. Sama reakcja nazywana jest łańcuchową, gdyż powtarzana w ściśle określonych cyklach pomnaża liczbę tworzonych kopii DNA w zawrotnym tempie. W każdym kolejnym cyklu reakcji PCR liczba kopii wybranego fragmentu DNA zwiększa się dwukrotnie. Jeżeli dysponujemy na przykład tylko jedną cząsteczkę DNA, na początku mamy tylko 2 kopie interesującej nas sekwencji, po jednej na każdej z komplementarnych nici DNA. Po pierwszym cyklu reakcji PCR mamy 4 kopie. Po drugim cyklu już 8. Po trzecim cyklu 16. W ten sposób liczba tworzonych kopii wybranej sekwencji DNA wzrasta wykładniczo i jest równa liczbie kopii startowych, podniesionej do potęgi, równej liczbie cykli reakcji PCR. Krótko mówiąc z początkowej jednej cząstki DNA zawierającej 2 kopie, po 20 cyklach otrzymamy około 1 miliona. To tak jakby szukając igły w stogu siana móc zmienić siano w stóg igieł. Taka magiczna sztuczka w celu ułatwienia poszukiwań. W naszym przypadku zamiast magii stosujemy sprytną reakcję PCR, efekt jest jednak równie spektakularny. Cały sekret metody PCR polega na tym, że aby zwielokrotnić odcinek DNA musimy znać sekwencje ok. 20 liter z każdej z strony wybranego do kopiowania fragmentu. Następnie dzięki specjalnemu urządzeniu do syntezy, tworzymy oligonukleotydy, czyli krótkie, jednoniciowe DNA. Zbudowane od nowa oligonukleotydy, muszą mieć sekwencję komplementarną, do każdego z wybranych, odcinków z obu stron interesującego nas fragmentu DNA. Tak stworzone dwa oligonukleotydy, w reakcji PCR nazywamy starterami. Komplementarność polega na tym, że każdej literze na nici DNA odpowiada zawsze inna, ściśle określona literka. Literze A odpowiada zawsze T, a C odnajduje zadowolenie wyłącznie w towarzystwie litery G. Dwie różne nici DNA mogą łączyć się ze sobą tworząc dwuniciową spiralę DNA, wyłącznie jeżeli sekwencje obu nici są względem siebie komplementarne.
Wnioski wyciągnięte przez Kary’ego Mullisa, podczas jazdy z Berkeley do Anderson Valley, okazały całkowicie prawidłowe. Dotyczyło to zarówno samego odkrycia jak i jego wagi oraz wpływu na rozwój badań genetycznych. Metoda opracowana przez Kary’ego stała się dla biologów molekularnych, tym czym było dla mikrobiologów udoskonalenie w XVII wieku mikroskopu przez Antoni van Leeuwenhoeka. Mikroskop pozwolił po raz pierwszy powiększyć i zbadać świat mikroorganizmów niewidocznych gołym okiem. Analogicznie metoda odkryta przez Kary’ego Mullisa pozwoliła na powiększenie liczby kopii dowolnego fragmentu DNA, do poziomu pozwalającego na swobodne badanie i odczytywanie informacji genetycznej. Metodzie opracowanej przez Kary’ego, nadano dość groźnie brzmiącą nazwę: Reakcja Łańcuchowa Polimerazy (Polymerase Chain Reaction), w skrócie PCR. Wbrew oczywistym skojarzeniom PCR nie ma nic wspólnego, z groźnym grzybem atomowym i tym czym zazwyczaj reakcja łańcuchowa kończy się w filmach z Bondem lub w Mission Impossible. W tym przypadku jest to zwykła zbieżność pojęć. Reakcja PCR, naśladuje naturalny proces kopiowania – replikacji DNA, pozwalając na zwielokrotnianie wybranego fragmentu informacji genetycznej (więcej na temat replikacji DNA znajdziecie we wpisie pt. “Genetyczny odcisk palca”). Zarówno w procesie naturalnej replikacji jak i w przypadku reakcji PCR enzymem odpowiedzialnym za szybkie i dokładne kopiowanie DNA jest polimeraza DNA. Sama reakcja nazywana jest łańcuchową, gdyż powtarzana w ściśle określonych cyklach pomnaża liczbę tworzonych kopii DNA w zawrotnym tempie. W każdym kolejnym cyklu reakcji PCR liczba kopii wybranego fragmentu DNA zwiększa się dwukrotnie. Jeżeli dysponujemy na przykład tylko jedną cząsteczkę DNA, na początku mamy tylko 2 kopie interesującej nas sekwencji, po jednej na każdej z komplementarnych nici DNA. Po pierwszym cyklu reakcji PCR mamy 4 kopie. Po drugim cyklu już 8. Po trzecim cyklu 16. W ten sposób liczba tworzonych kopii wybranej sekwencji DNA wzrasta wykładniczo i jest równa liczbie kopii startowych, podniesionej do potęgi, równej liczbie cykli reakcji PCR. Krótko mówiąc z początkowej jednej cząstki DNA zawierającej 2 kopie, po 20 cyklach otrzymamy około 1 miliona. To tak jakby szukając igły w stogu siana móc zmienić siano w stóg igieł. Taka magiczna sztuczka w celu ułatwienia poszukiwań. W naszym przypadku zamiast magii stosujemy sprytną reakcję PCR, efekt jest jednak równie spektakularny. Cały sekret metody PCR polega na tym, że aby zwielokrotnić odcinek DNA musimy znać sekwencje ok. 20 liter z każdej z strony wybranego do kopiowania fragmentu. Następnie dzięki specjalnemu urządzeniu do syntezy, tworzymy oligonukleotydy, czyli krótkie, jednoniciowe DNA. Zbudowane od nowa oligonukleotydy, muszą mieć sekwencję komplementarną, do każdego z wybranych, odcinków z obu stron interesującego nas fragmentu DNA. Tak stworzone dwa oligonukleotydy, w reakcji PCR nazywamy starterami. Komplementarność polega na tym, że każdej literze na nici DNA odpowiada zawsze inna, ściśle określona literka. Literze A odpowiada zawsze T, a C odnajduje zadowolenie wyłącznie w towarzystwie litery G. Dwie różne nici DNA mogą łączyć się ze sobą tworząc dwuniciową spiralę DNA, wyłącznie jeżeli sekwencje obu nici są względem siebie komplementarne.
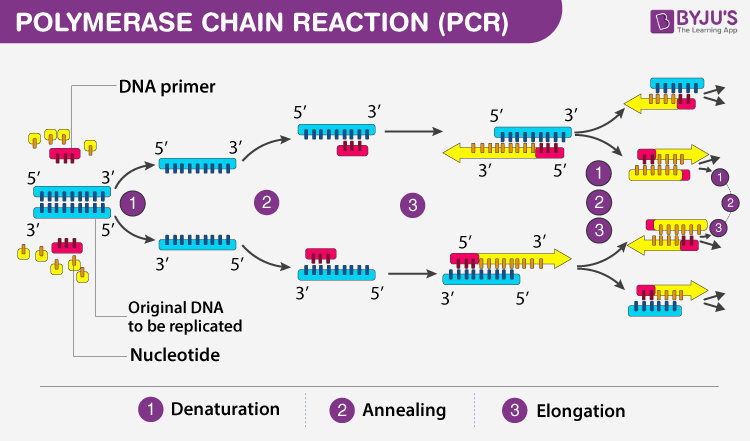
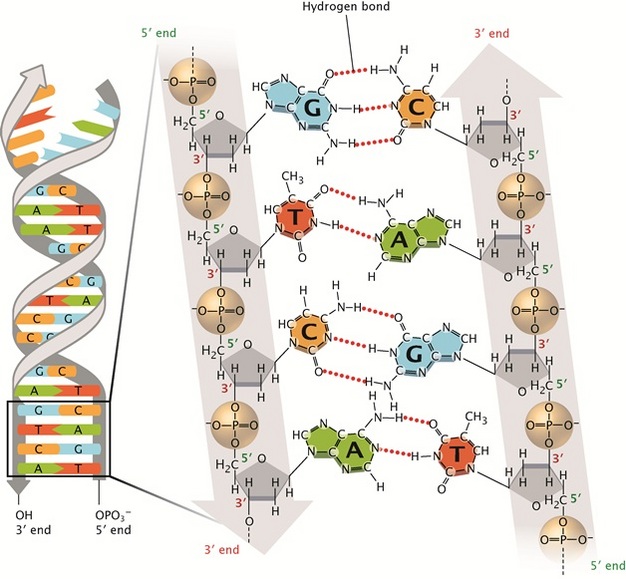 Spirala DNA, nazywana również całkiem zgrabnie helisą, ma pewną specyficzną właściwość, którą wykorzystuje metoda PCR. W podwyższonej temperaturze rozplata się tworząc dwie osobne nici DNA. Proces ten nazywamy denaturacją. Pomimo naturalnych i oczywistych skojarzeń, zjawisko to nie ma jednak nic wspólnego z pewnym fioletowym trunkiem, dość popularnym wśród mało wybrednych miłośników napojów alkoholowych. Po prostu podgrzana do ok. 95C helisa DNA dzieli się z powrotem na dwie komplementarne, niezależne nici. Aby przeprowadzić reakcję PCR do mieszaniny reakcyjnej dodawane są syntetyczne startery o sekwencji komplementarnej do fragmentów na obu końcach kopiowanego odcinka DNA. Oprócz starterów do mieszaniny dodawana jest również polimeraza DNA oraz cegiełki budulca, z którego tworzony jest DNA tzw. nukleotydy. Ponieważ w trakcie denaturacji trzeba wszystko podgrzać do ok. 95C biolodzy molekularni wykorzystali specjalną polimerazę DNA, odporną na wysokie temperatury. Najczęściej jest to tzw. polimeraza Taq. To prawdziwy twardziel wśród polimeraz DNA, wyizolowany z bakterii o dość egzotycznej nazwie Thermus aquaticus żyjących w gorących źródłach. Polimeraza Taq nie tylko bez kłopotu wytrzymuje podgrzanie w prawie wrzącej mieszaninie reakcyjnej, potrafi również pracować i doskonale kopiować DNA w wysokiej temperaturze. Następnym krokiem w sprytnej reakcji PCR jest obniżenie temperatury do ok. 45-60C. W niskiej temperaturze jednoniciowy DNA zaczyna czuć wyraźną potrzebę towarzystwa i łączy się ponownie z komplementarną nicią. Jednak w tym przypadku w mieszaninie są już startery. Nie jest to jednak jeden lub kilka starterów, ale miliony syntetycznych, komplementarnych kopii ok. 20 literowych fragmentów z każdego końca wybranej sekwencji. Nic dziwnego, że DNA, który chcemy skopiować nie łączy się z powrotem z drugą, całkowitą nicią DNA, ale z naszymi starterami. W trzecim etapie reakcji PCR mieszanina jest znów podgrzewana, tym razem do 72C. W tej temperaturze ciepłolubna polimeraza Taq czuje się najlepiej i z zapałem zaczyna wydłużać startery kopiując przy okazji wyjściowy, całkowity odcinek DNA. Następnie cały cykl: denaturacji, przyłączania starterów oraz ich wydłużania powtarzany jest wielokrotnie dzięki odpowiedniemu podgrzewaniu i oziębianiu mieszaniny reakcyjnej. Metoda jest prosta i łatwo ją zautomatyzować. W każdym cyklu liczba kopii naszego wybranego fragmentu DNA wzrasta wykładniczo. Proste, tanie, eleganckie. Zupełnie jak z reklamy posezonowej wyprzedaży sukienek w H&M.
Spirala DNA, nazywana również całkiem zgrabnie helisą, ma pewną specyficzną właściwość, którą wykorzystuje metoda PCR. W podwyższonej temperaturze rozplata się tworząc dwie osobne nici DNA. Proces ten nazywamy denaturacją. Pomimo naturalnych i oczywistych skojarzeń, zjawisko to nie ma jednak nic wspólnego z pewnym fioletowym trunkiem, dość popularnym wśród mało wybrednych miłośników napojów alkoholowych. Po prostu podgrzana do ok. 95C helisa DNA dzieli się z powrotem na dwie komplementarne, niezależne nici. Aby przeprowadzić reakcję PCR do mieszaniny reakcyjnej dodawane są syntetyczne startery o sekwencji komplementarnej do fragmentów na obu końcach kopiowanego odcinka DNA. Oprócz starterów do mieszaniny dodawana jest również polimeraza DNA oraz cegiełki budulca, z którego tworzony jest DNA tzw. nukleotydy. Ponieważ w trakcie denaturacji trzeba wszystko podgrzać do ok. 95C biolodzy molekularni wykorzystali specjalną polimerazę DNA, odporną na wysokie temperatury. Najczęściej jest to tzw. polimeraza Taq. To prawdziwy twardziel wśród polimeraz DNA, wyizolowany z bakterii o dość egzotycznej nazwie Thermus aquaticus żyjących w gorących źródłach. Polimeraza Taq nie tylko bez kłopotu wytrzymuje podgrzanie w prawie wrzącej mieszaninie reakcyjnej, potrafi również pracować i doskonale kopiować DNA w wysokiej temperaturze. Następnym krokiem w sprytnej reakcji PCR jest obniżenie temperatury do ok. 45-60C. W niskiej temperaturze jednoniciowy DNA zaczyna czuć wyraźną potrzebę towarzystwa i łączy się ponownie z komplementarną nicią. Jednak w tym przypadku w mieszaninie są już startery. Nie jest to jednak jeden lub kilka starterów, ale miliony syntetycznych, komplementarnych kopii ok. 20 literowych fragmentów z każdego końca wybranej sekwencji. Nic dziwnego, że DNA, który chcemy skopiować nie łączy się z powrotem z drugą, całkowitą nicią DNA, ale z naszymi starterami. W trzecim etapie reakcji PCR mieszanina jest znów podgrzewana, tym razem do 72C. W tej temperaturze ciepłolubna polimeraza Taq czuje się najlepiej i z zapałem zaczyna wydłużać startery kopiując przy okazji wyjściowy, całkowity odcinek DNA. Następnie cały cykl: denaturacji, przyłączania starterów oraz ich wydłużania powtarzany jest wielokrotnie dzięki odpowiedniemu podgrzewaniu i oziębianiu mieszaniny reakcyjnej. Metoda jest prosta i łatwo ją zautomatyzować. W każdym cyklu liczba kopii naszego wybranego fragmentu DNA wzrasta wykładniczo. Proste, tanie, eleganckie. Zupełnie jak z reklamy posezonowej wyprzedaży sukienek w H&M.
 Dziś PCR jest podstawową techniką badawczą stosowaną biologii molekularnej. Przeprowadzaniem reakcji zajmują się w pełni automatyczne urządzenia do namnażania DNA nazywane termocyklerami. Nie jest zaskoczeniem, że nasz Kary Mullis otrzymał za opracowanie metody PCR w 1993 r. Nagrodę Nobla w dziedzinie chemii. Sława oraz prestiż związany z dołączeniem do szacownego grona noblistów trochę nadwyrężyły niestety niezbyt silną konstrukcję psychiczną Kary’ego. Porzucił on na pewien czas pracę badawczą na rzecz głoszenia różnych teorii spiskowych oraz walkę z dowodami na rzecz wirusowego pochodzenie AIDS. Ponadto zajął się również promowaniem astrologii, oraz założył własną firmę o dumnej nazwie: Star Gene (Geny Gwiazd). Firma miała zajmować się produkcją biżuterii zawierającej namnoży DNA sławnych ludzi: aktorów, sportowców, gwiazd rocka np.: Marilyn Monroe czy też Elvisa Presleya. Niestety ku rozczarowaniu Kary’ego akurat ten pomysł nie przyniósł mu zbyt wiele sławy i bogactwa.
Dziś PCR jest podstawową techniką badawczą stosowaną biologii molekularnej. Przeprowadzaniem reakcji zajmują się w pełni automatyczne urządzenia do namnażania DNA nazywane termocyklerami. Nie jest zaskoczeniem, że nasz Kary Mullis otrzymał za opracowanie metody PCR w 1993 r. Nagrodę Nobla w dziedzinie chemii. Sława oraz prestiż związany z dołączeniem do szacownego grona noblistów trochę nadwyrężyły niestety niezbyt silną konstrukcję psychiczną Kary’ego. Porzucił on na pewien czas pracę badawczą na rzecz głoszenia różnych teorii spiskowych oraz walkę z dowodami na rzecz wirusowego pochodzenie AIDS. Ponadto zajął się również promowaniem astrologii, oraz założył własną firmę o dumnej nazwie: Star Gene (Geny Gwiazd). Firma miała zajmować się produkcją biżuterii zawierającej namnoży DNA sławnych ludzi: aktorów, sportowców, gwiazd rocka np.: Marilyn Monroe czy też Elvisa Presleya. Niestety ku rozczarowaniu Kary’ego akurat ten pomysł nie przyniósł mu zbyt wiele sławy i bogactwa.
 Pomimo wspomnianych słabości emocjonalnych swego odkrywcy, wkład jaki metoda PCR wniosła w rozwój badań genetycznych nie pozostawia najmniejszych wątpliwości. PCR jest obecnie podstawową techniką stosowaną podczas badania DNA. W idealnie precyzyjny sposób pozwala ona wybrać i następnie namnożyć dowolny fragment genomu. Następnie namnożony do odpowiedniej ilości odcinek DNA można np.: odczytać by ustalić jego informację genetyczną lub wprowadzić do innego organizmu, w celu dokonania modyfikacji genetycznej. Metoda PCR zrewolucjonizowała nie tylko badania DNA prowadzona w placówkach naukowych. Stała się również podstawowym narzędziem wykorzystywanym na co dzień przez wiele instytucji, których o związek genetycznymi metodami badawczymi nigdy byśmy nie podejrzewali. Reakcja PCR jest powszechnie stosowana przez: policję, inspekcję sanitarną i weterynaryjną, służbę zdrowia oraz sądownictwo. Wystarczy wymienić tylko kilka z bardziej popularnych, praktycznych sposobów wykorzystania metody PCR:
Pomimo wspomnianych słabości emocjonalnych swego odkrywcy, wkład jaki metoda PCR wniosła w rozwój badań genetycznych nie pozostawia najmniejszych wątpliwości. PCR jest obecnie podstawową techniką stosowaną podczas badania DNA. W idealnie precyzyjny sposób pozwala ona wybrać i następnie namnożyć dowolny fragment genomu. Następnie namnożony do odpowiedniej ilości odcinek DNA można np.: odczytać by ustalić jego informację genetyczną lub wprowadzić do innego organizmu, w celu dokonania modyfikacji genetycznej. Metoda PCR zrewolucjonizowała nie tylko badania DNA prowadzona w placówkach naukowych. Stała się również podstawowym narzędziem wykorzystywanym na co dzień przez wiele instytucji, których o związek genetycznymi metodami badawczymi nigdy byśmy nie podejrzewali. Reakcja PCR jest powszechnie stosowana przez: policję, inspekcję sanitarną i weterynaryjną, służbę zdrowia oraz sądownictwo. Wystarczy wymienić tylko kilka z bardziej popularnych, praktycznych sposobów wykorzystania metody PCR:
- Namnażanie śladowych ilości DNA, pozostawionych na miejscu przestępstwa w celu identyfikacji sprawcy.
- Testy genetyczne pozwalające przewidywać skłonność do chorób genetycznych.
- Testy na ojcostwo.
- Ocena skażenia żywności szkodliwymi mikroorganizm takimi jak np.: Salmonella.
- Identyfikacja obecności GMO (organizmów modyfikowanych genetycznie).
- Błyskawiczne wykrywanie rodzaju użytej broni biologicznej np.: wąglika, podczas działań wojennych lub w przypadku ataku terrorystycznego.
- Określanie gatunku ryby, z której pochodzi kawior, w celu wykrycia zafałszowania bardzo drogiego kawioru z jesiotra, kawiorem tańszych gatunków.
- Namnażanie DNA wymarłych organizmów np.: mamuta, w celu odczytania informacji genetycznej oraz być może w przyszłości przywrócenia ich do życia.
 Metoda PCR doczekała się wielu modyfikacji i ulepszeń. Jednym z bardziej popularnych sposobów rozwinięcia możliwości, które daje reakcja błyskawicznego namnażanie DNA jest tzw. PCR w czasie rzeczywistym (real-time PCR). Real-time PCR wykorzystuje opisany wcześniej mechanizm kopiowania DNA, do którego wprowadzono jednak kilka istotnych z ulepszeń. Dość ważną przewagą real-time PCR nad metodą klasyczną, jest możliwość oceny ilości namnożonych kopii DNA już w trakcie trwania reakcji. Wcześniej trzeba było doprowadzić cały proces do końca i dopiero wtedy wykonywać dalszą ocenę np.: sprawdzić czy reakcja zaszła, czy namnożony fragment DNA ma przewidywaną długość itd. Wymagało to sporo dodatkowej pracy oraz czasu. Wyobraźcie sobie biednego laboranta, który o 15:00 otrzymał zlecenie zbadania pilnych próbek przy użyciu metody PCR. Niestety tego dnia o 16:00 zaczyna się Liga Mistrzów, a on nie zdążył na jeszcze nawet kupić piwa. Stosując klasyczną metodę PCR nasz biedny laborant prawdopodobnie spóźniłby się nawet na dogrywkę. Stosując real-time PCR nie tylko skończy wszystkie badania przed meczem, ale również kupi piwo i wyśle posta do kolegów kibiców na Facebooku.
Metoda PCR doczekała się wielu modyfikacji i ulepszeń. Jednym z bardziej popularnych sposobów rozwinięcia możliwości, które daje reakcja błyskawicznego namnażanie DNA jest tzw. PCR w czasie rzeczywistym (real-time PCR). Real-time PCR wykorzystuje opisany wcześniej mechanizm kopiowania DNA, do którego wprowadzono jednak kilka istotnych z ulepszeń. Dość ważną przewagą real-time PCR nad metodą klasyczną, jest możliwość oceny ilości namnożonych kopii DNA już w trakcie trwania reakcji. Wcześniej trzeba było doprowadzić cały proces do końca i dopiero wtedy wykonywać dalszą ocenę np.: sprawdzić czy reakcja zaszła, czy namnożony fragment DNA ma przewidywaną długość itd. Wymagało to sporo dodatkowej pracy oraz czasu. Wyobraźcie sobie biednego laboranta, który o 15:00 otrzymał zlecenie zbadania pilnych próbek przy użyciu metody PCR. Niestety tego dnia o 16:00 zaczyna się Liga Mistrzów, a on nie zdążył na jeszcze nawet kupić piwa. Stosując klasyczną metodę PCR nasz biedny laborant prawdopodobnie spóźniłby się nawet na dogrywkę. Stosując real-time PCR nie tylko skończy wszystkie badania przed meczem, ale również kupi piwo i wyśle posta do kolegów kibiców na Facebooku.
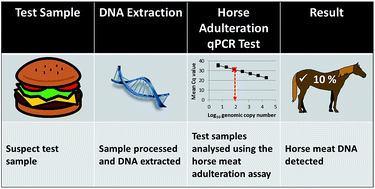
 Sprytne udoskonalenie standardowej metody PCR, polega na wykorzystaniu znaczników fluorescencyjnych do badania reakcji w czasie rzeczywistym. Znaczniki fluorescencyjne to substancje, które pod wpływem wzbudzenia przez promieniowania elektromagnetyczne emitują światło. Brzmi dość mało przyjaźnie, ale jest całkiem proste i przyjemne. Zasada jest taka sama jak na każdym Fluo Glow Party w podczas sobotniego klubingu. Malujemy się farbą fluorescencyjną, a DJ sprawia, że świecimy pod wpływem “promieniowania elektromagnetycznego” z lampy UV. W przypadku reakcji real-time PCR podczas kopiowania DNA ilość aktywnego znacznika fluorescencyjnego wzrasta proporcjonalnie do ilości namnożonego DNA. Dzięki temu oceniając poziom fluorescencji możemy mierzyć poziom ilość kopiowanego DNA już podczas trwania reakcji PCR. Sprytne i skuteczne. Nasza imprezowy real-time PCR ma jeszcze jedną zaletę. Dzięki pomiarowi szybkości zachodzącej reakcji możemy również ocenić początkową ilość badanego fragmentu DNA. Jest to bardzo użyteczne, gdy chcemy np.: poznać stopień skażenia badanej próbki chorobotwórczymi mikroorganizmami. Dzięki prawie całkowitej automatyzacji testów, oraz wspomnianej możliwości oceny początkowej ilości badanego DNA, real-time PCR jest dziś podstawową techniką stosowaną w celu genetycznej identyfikacji różnych organizmów. Metoda ta jest standardowo wykorzystywana do szybkiej identyfikacji oraz oceny poziomu patogenów, takich jak: bakterie, wirusy oraz grzyby. Real-time PCR jest również z powodzeniem stosowany w celu wykrywanie oszustw. Metoda ta była w 2013 roku cichym bohaterem, śledztwa dotyczącego afery z potajemnym dodawania koniny do przetworów mięsnych. Dzięki przeprowadzonym badaniom okazało się, że pewna część mielonej wołowiny sprzedawanej w wielu krajach Europy, w przeszłości częściej rżała niż muczała.
Sprytne udoskonalenie standardowej metody PCR, polega na wykorzystaniu znaczników fluorescencyjnych do badania reakcji w czasie rzeczywistym. Znaczniki fluorescencyjne to substancje, które pod wpływem wzbudzenia przez promieniowania elektromagnetyczne emitują światło. Brzmi dość mało przyjaźnie, ale jest całkiem proste i przyjemne. Zasada jest taka sama jak na każdym Fluo Glow Party w podczas sobotniego klubingu. Malujemy się farbą fluorescencyjną, a DJ sprawia, że świecimy pod wpływem “promieniowania elektromagnetycznego” z lampy UV. W przypadku reakcji real-time PCR podczas kopiowania DNA ilość aktywnego znacznika fluorescencyjnego wzrasta proporcjonalnie do ilości namnożonego DNA. Dzięki temu oceniając poziom fluorescencji możemy mierzyć poziom ilość kopiowanego DNA już podczas trwania reakcji PCR. Sprytne i skuteczne. Nasza imprezowy real-time PCR ma jeszcze jedną zaletę. Dzięki pomiarowi szybkości zachodzącej reakcji możemy również ocenić początkową ilość badanego fragmentu DNA. Jest to bardzo użyteczne, gdy chcemy np.: poznać stopień skażenia badanej próbki chorobotwórczymi mikroorganizmami. Dzięki prawie całkowitej automatyzacji testów, oraz wspomnianej możliwości oceny początkowej ilości badanego DNA, real-time PCR jest dziś podstawową techniką stosowaną w celu genetycznej identyfikacji różnych organizmów. Metoda ta jest standardowo wykorzystywana do szybkiej identyfikacji oraz oceny poziomu patogenów, takich jak: bakterie, wirusy oraz grzyby. Real-time PCR jest również z powodzeniem stosowany w celu wykrywanie oszustw. Metoda ta była w 2013 roku cichym bohaterem, śledztwa dotyczącego afery z potajemnym dodawania koniny do przetworów mięsnych. Dzięki przeprowadzonym badaniom okazało się, że pewna część mielonej wołowiny sprzedawanej w wielu krajach Europy, w przeszłości częściej rżała niż muczała.
 Metoda PCR zmieniła gruntownie sposób prowadzenia badań DNA, nie tylko upraszczając je, ale dając nowe możliwości, o których wcześniej można było jedynie marzyć. Bez wielkiej przesady można powiedzieć, że bez metody PCR nie byłaby możliwa obecna biotechnologiczna rewolucja, która zmieniła całkowicie niektóre obszary globalnej gospodarki. Dzięki łatwej możliwości namnażania wybranych odcinków DNA stało się możliwe dokonanie przełomowych odkryć dotyczących np.: opracowania i wytwarzania nowych lekarstw, uprawy nowych odmian roślin i zwierząt, czy też biotechnologicznej syntezy niektórych substancji chemicznych np.: witamin. Wszystko to zaczęło się jednak od przebłysku geniuszu Kary’ego Mullisa oraz niedocenianej umiejętności zadawania fundamentalnego pytania „co by było gdyby…” Sam Kary dodał w wywiadzie opublikowanym w California Monthly, że w uzyskaniu właściwej odpowiedzi pomogło mu również spore doświadczenie w zażywaniu LSD. Substancja jak stwierdził Kary rozwinęła jego umysł bardziej niż jakiekolwiek wykłady na uniwersytecie. Można by stąd wyciągnąć dość kontrowersyjny wniosek, że kandydat na genialnego biochemika, niekoniecznie musi być pilnym studentem, który nie pije, nie pali i nigdy w życiu nic nie brał. Jedno i drugiego zupełnie się nie wyklucza, a zgodnie z opinią Kary’ego często nawet wzajemnie się wspiera. Warto jednak dodać, że Kary twierdził również, że w 1985 r. spotkał koło swojej chatki w lesie świecącego na zielono szopa pracza, który w dodatku grzecznie się z nim przywitał. Biorąc pod uwagę tą historię, jak i wspomniane wcześniej poglądy Kary’ego, szczerze wątpię aby naszego noblistę, warto uznać za osobę mogącą być autorytetem w sprawach istotnych życiowych wyborów. Włączając w to decyzję dotyczącą sposobu rozwoju naszego umysłu.
Metoda PCR zmieniła gruntownie sposób prowadzenia badań DNA, nie tylko upraszczając je, ale dając nowe możliwości, o których wcześniej można było jedynie marzyć. Bez wielkiej przesady można powiedzieć, że bez metody PCR nie byłaby możliwa obecna biotechnologiczna rewolucja, która zmieniła całkowicie niektóre obszary globalnej gospodarki. Dzięki łatwej możliwości namnażania wybranych odcinków DNA stało się możliwe dokonanie przełomowych odkryć dotyczących np.: opracowania i wytwarzania nowych lekarstw, uprawy nowych odmian roślin i zwierząt, czy też biotechnologicznej syntezy niektórych substancji chemicznych np.: witamin. Wszystko to zaczęło się jednak od przebłysku geniuszu Kary’ego Mullisa oraz niedocenianej umiejętności zadawania fundamentalnego pytania „co by było gdyby…” Sam Kary dodał w wywiadzie opublikowanym w California Monthly, że w uzyskaniu właściwej odpowiedzi pomogło mu również spore doświadczenie w zażywaniu LSD. Substancja jak stwierdził Kary rozwinęła jego umysł bardziej niż jakiekolwiek wykłady na uniwersytecie. Można by stąd wyciągnąć dość kontrowersyjny wniosek, że kandydat na genialnego biochemika, niekoniecznie musi być pilnym studentem, który nie pije, nie pali i nigdy w życiu nic nie brał. Jedno i drugiego zupełnie się nie wyklucza, a zgodnie z opinią Kary’ego często nawet wzajemnie się wspiera. Warto jednak dodać, że Kary twierdził również, że w 1985 r. spotkał koło swojej chatki w lesie świecącego na zielono szopa pracza, który w dodatku grzecznie się z nim przywitał. Biorąc pod uwagę tą historię, jak i wspomniane wcześniej poglądy Kary’ego, szczerze wątpię aby naszego noblistę, warto uznać za osobę mogącą być autorytetem w sprawach istotnych życiowych wyborów. Włączając w to decyzję dotyczącą sposobu rozwoju naszego umysłu.
Zdjęcia źródło:
- https://www.thoughtco.com/dna-replication-3981005
- https://dranshublog.com/the-story-of-kary-mullis-and-the-invention-of-pcr/
- https://www.nature.com/scitable/topicpage/discovery-of-dna-structure-and-function-wa
- https://byjus.com/biology/recombinant-dna-technology-process/
- https://biosistemika.com/blog/qpcr-technology-basics/
- https://www.amazon.com/Dancing-Naked-Mind-Field-Mullis/dp/0679774009

